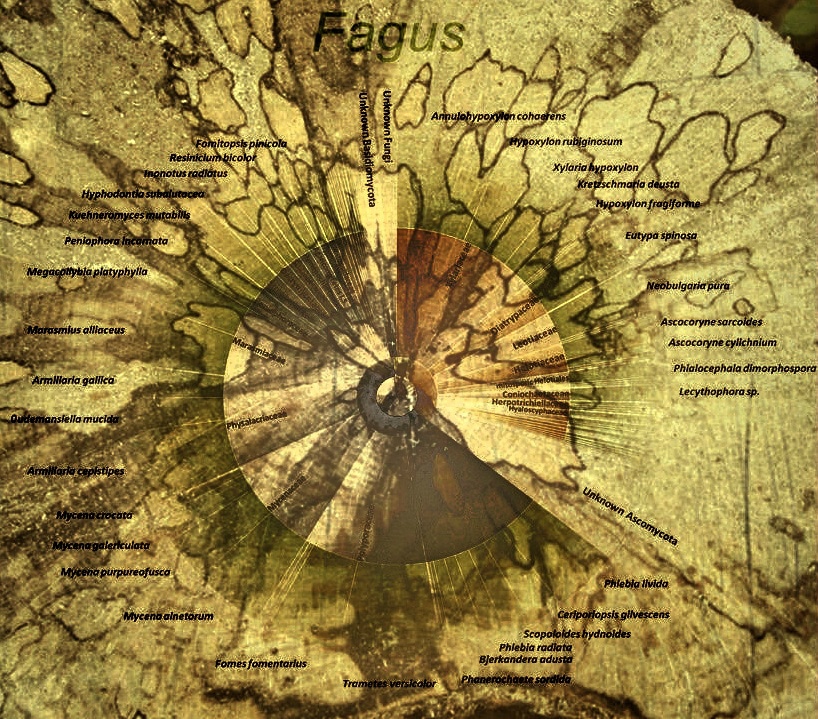
BELongDead – Pilzliche Funktionelle Diversität – Halle/Zittau/Grafenau Strukturelle Diversität von Pilzen und Bakterien im Totholz und die Expression von holzabbaurelevanten Genen
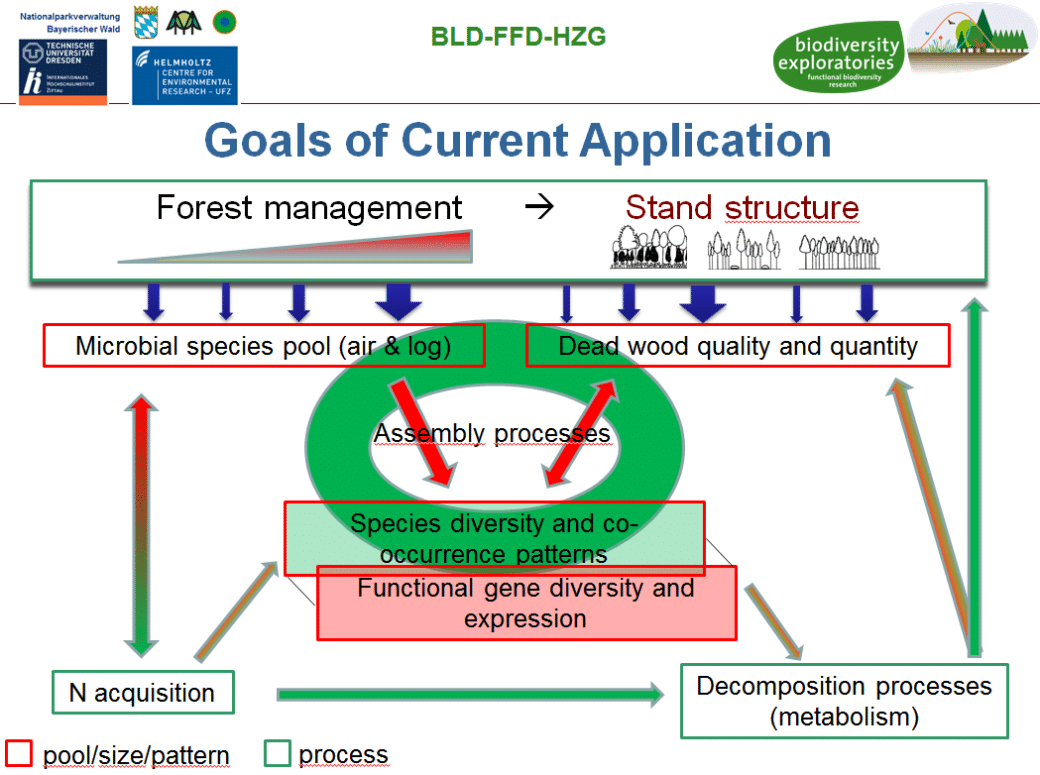
Im Vergleich zu den Vorgängerprojekten FunWood I-II, die sich weitestgehend mit dem Einfluss von Waldbewirtschaftung auf mikrobielle Diversität im bereits vorhandenem, in Zersetzung befindlichem Totholz unter Feldbedingungen beschäftigt hat, verlagert sich nunmehr unser Fokus auf eine experimentelle Plattform. Das BELongDead Experiment wurde 2008 unter der Federführung von Prof. Dr. E.D. Schulze (MPI Biogeochemie Jena) initialisiert, mit dem Ziel den Einfluss des umliegenden Habitats auf Totholz und dessen Abbauprozesse zu untersuchen. Ein weiterer Schwerpunkt liegt hierbei auf der Langzeitbeobachtung der Besiedlung der Totholzstämme durch verschiedenste Organismen.
Es soll untersucht werden, inwiefern I) das umgebende Ökosystem den Totholzabbau beeinflusst, II) wie die Totholzbesiedlung erfolgt und III) wie Mikroorganismen den Totholzabbau steuern und damit Ökosystemprozesse wie z.B. den Nährstoffumsatz beeinflussen. BELongDead ermöglicht uns den Einfluss von Landnutzung in Form von Waldbewirtschaftung an einem standardisiertem Satz Totholz (12+1 verschiedene Baumarten von gleicher Größe und gleichem Zersetungsbeginn), die gleichmäßig verteilt in 3 Replikaten in den 3 Exploratorien sind und in jeweils 3×3 verschieden bewirtschafteten Plots, zu untersuchen.
Ziel unseres Projektes ist es, modernste molekularbiologische Methoden mit klassischen Fruchtkörperkartierungen und Sporensammlungen zu kombinieren, um I) Art und Quantität der Holzabbaus und verschieden Waldbewirtschaftungsaspekten zu determinieren, II) Verbreitungs- und Sukzessionsmuster von Pilzen über einen längeren Zeitraum zu beobachten und zu untersuchen, III) Pilzaktivität auf Transkriptom- und Enzymebene zu bestimmen und diese Ergebnisse mit den Prozessdaten verweben, IV) daraus resultierende Veränderungen in der Holzchemie bestimmen, V) den Einfluss von N-fixierenden Bakterien auf die Diversität von Pilzen bestimmen und vi) letztendlich Schlüsselarten in diesen komplexen Prozessen zu identifizieren.
1. Erhöhte Waldbewirtschaftungsintensität reduziert den Artenpool holzbewohnender Pilze auf Landschafts- und Waldbestandsebene.
2. Intensive Waldbewirtschaftung ist ein Habitatfilter, der bestimmte Arten mit bestimmten Lebensstrategien (z.B. Generalisten) begünstigt.
3. Waldbewirtschaftung entspannt kompetitive Interaktionen zwischen holzbewohnenden Pilzen, was zu höheren Holzzersetzungsraten führt.
4. Holzzersetzungsprozesse sind anhand von Fruchtkörperdatierungen, kombiniert mit molekular erhobenen Daten zu phylogenetischer und funktioneller Diversität sehr gut vorhersagbar.
5. Es gibt beständige mikrobielle N-Fixierung im Totholz, und bestimmte Bakterien treten nicht-zufällig mit bestimmten Pilzen auf.
6. Unter den typischen von Pilzen exprimierten Peroxidasen (Class II Peroxidasen, Hem-Thiolat Peroxidasen) erwarten wir, dass Manganperoxidasen am diversesten und abundantesten von allen lignin-modifizierenden Enzymen sind.
7. Peroxidasen-Transkriptom Diversität erhöht sich mit größerer Diversität von Weißfäulepilzen.
Zur Beantwortung unserer Fragestellungen verwenden wir modernste molekularbiologische Techniken auf DNA und RNA-Ebene, darunter auch sog. „Next Generation Sequencing“-Techniken (NGS). Des Weiteren wollen wir die tatsächliche N-Fixierung im Holz nachweisen und die Allokation des N den entsprechenden Beteiligten zuordnen. Hierfür planen wir ein SIP Experiment („stable isotope probing“), zusammen mit NGS Metagenomik, real-time PCR und Bioanalytik (Acetylen-Reduktions-Tests, Massenspektrographie).
Neben intensiven Fruchtkörperkartierungen werden ebenfalls Sporenkollektoren installiert, um luftsrömungsbasierte Verbreitungsmuster zu identifizieren, und zum anderen vergleichen zu können, welche Pilze potentiell vorkommen (species pool), und welche bereits im Totholz angesiedelt sind.